добавить сегменты к точечной диаграмме
(это следуетggplot2 loess Q за что я получил хороший ответ) - ведущий к этому сюжету
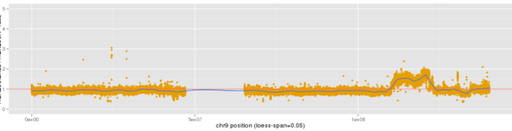
Мои знания R весьма ограничены (извините!)
Я строю график разброса, используя данные из таблицы data1.
<code>data1<-NaRV.omit(data[,c(2,3,7,10)]) #(2=start, 3=end, 7=value, 10=type) ylabs='E / A - ratio' p1<-ggplot(data1, aes(x=start, y=value)) + ylim(0,5) + geom_point(shape=points, col=pointcol1, na.rm=T) + geom_hline(aes(yintercept=1, col=linecol)) + geom_smooth(method="loess", span=spanv, fullrange=F, se=T, na.rm=T) + # xlab(xlabs) + ylab(ylabs) </code>
В некоторых регионах нет данных (в том числе одна большая область посередине, но есть и более мелкие дискретные области), где я хотел бы нарисовать цветные сегменты при y = 0, чтобы проиллюстрировать этот факт
Я объединил оба типа данных в одну таблицу со столбцом метки # 10 = «тип» (содержимое для данных разброса = "cnv" и для данных без данных = "nregion"). у nregions есть 0 в столбце значений.
Как я могу взять только «cnv»? данные для разброса и только «nregion»; данные для рисования сегментов; оба на одном участке?
Я нашел geom_segment:
<code>+ geom_segment(aes(x=data1$start, y=0, xend=data1$end, yend=0)) </code>
НО я не нашел способ подмножества для каждого подзаговора ggplot.
Спасибо
#### follow up on @gauden solutionПривет @gauden Я попробовал ваш подход, и это отчасти сработало. Моя проблема в том, что я не могу разделить мои данные так же хорошо, как вы используете] -1; 0], потому что мои nregions разбросаны (представлены синими точками и линиями на рисунке) и отличаются для каждого нового графика, как на этом изображении:
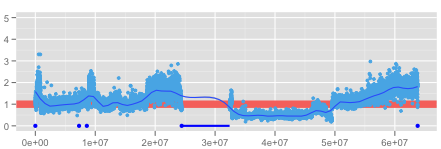
Следовательно, лесса, как и прежде, проходит через большой регион. Как я могу предотвратить лесс в nregions?
<code>############################# ## plot settings (edit below) spanv<-0.1 pointcol1="#E69F00" pointcol2="#56B4E9" pointcol3="#009E73" points=20 onecol="green" colnreg="blue" xlabs=paste(onechr, " position", " (loess-span=", spanv, ")", sep="") ##### end edit ############## ######################################################## ## using the center coordinate of each segment and points ## prepare plot #1 # plot E / A - ratio ## draw loess average for cnv ## draw line for nregion ylabs='E / A - ratio' p1<-ggplot(chrdata, aes(x=start+1000, y=E.R, group=type, label=type)) + ylim(0,5) + geom_hline(aes(yintercept=1, col=onecol)) + geom_point(data = chrdata[chrdata$type != 'nregion',], shape=points, col=pointcol2) + geom_smooth(data = chrdata[chrdata$type != 'nregion',], method="loess", span=spanv) + geom_point(data = chrdata[chrdata$type == 'nregion',], col=colnreg) + geom_segment(data = chrdata[chrdata$type == 'nregion',], aes(x=start, y=E.R, xend=end, yend=E.R), colour=colnreg, linetype=1, size=1) + xlab(xlabs) + ylab(ylabs) </code>