Эффективный расчет адаптированного к границе среднего значения окрестности
У меня есть изображение со значениями от 0 до 1. Мне нравится делать простое усреднение.
Но, более конкретно, для ячейки на границе изображения я бы хотел вычислить среднее значение пикселей для той части окрестности / ядра, которая находится в пределах изображения. Фактически это сводится к тому, чтобы адаптировать знаменатель «средней формулы», к числу пикселей, на которое вы делите сумму.
Мне удалось сделать это, как показано ниже сscipy.ndimage.generic_filter, но это далеко не по времени.
def fnc(buffer, count):
n = float(sum(buffer < 2.0))
sum = sum(buffer) - ((count - b) * 2.0)
return (sum / n)
avg = scipy.ndimage.generic_filter(image, fnc, footprint = kernel, \
mode = 'constant', cval = 2.0, \
extra_keywords = {'count': countkernel})
Details
kernel = square array (circle represented by ones)
Padding with 2's and not by zeroes since then I could not properly separate zeroes of the padded area and zeroes of the actual raster
countkernel = number of ones in the kernel
n = number of cells that lie within image by excluding the cells of the padded area identified by values of 2
Correct the sum by subtracting (number of padded cells * 2.0) from the original neighbourhood total sum
Update(s)
1) Заполнение NaN увеличивает расчет примерно на 30%:
def fnc(buffer):
return (numpy.nansum(buffer) / numpy.sum([~numpy.isnan(buffer)]))
avg = scipy.ndimage.generic_filter(image, fnc, footprint = kernel, \
mode = 'constant', cval = float(numpy.nan)
2) Применение решения, предложенногоYves Daoust (принятый ответ), безусловно, сокращает время обработки до минимума:
def fnc(buffer):
return numpy.sum(buffer)
sumbigimage = scipy.ndimage.generic_filter(image, fnc, \
footprint = kernel, \
mode = 'constant', \
cval = 0.0)
summask = scipy.ndimage.generic_filter(mask, fnc, \
footprint = kernel, \
mode = 'constant', \
cval = 0.0)
avg = sumbigimage / summask
3) Опираясь наYves' Совет использовать дополнительный двоичный образ, который на самом деле применяется маска, я наткнулся на принципмаскированные массивы, Таким образом, только один массив должен быть обработан, потому что маскированный массив "blends" изображение и маска вместе.
Небольшая деталь о массиве маски: вместо заполнения внутренней части (экстента исходного изображения) 1 и заполнения внешней части (границы) 0, как это было сделано в предыдущем обновлении, вы должны сделать наоборот. 1 в замаскированном массиве означает «неверный», 0 означает «действительный».
Этот код даже на 50% быстрее, чем код, представленный в обновлении 2):
maskedimg = numpy.ma.masked_array(imgarray, mask = maskarray)
def fnc(buffer):
return numpy.mean(buffer)
avg = scipy.ndimage.generic_filter(maskedimg, fnc, footprint = kernel, \
mode = 'constant', cval = 0.0)
--> I must correct myself here!
Я должен ошибаться во время проверки, так как после некоторых вычислений казалось, чтоscipy.ndimage.<filters> не может обрабатывать masked_arrays в том смысле, что во время операции фильтрации маска не учитывается.
Некоторые другие люди упоминали это тоже, например,Вот а такжеВот.
Сила изображения ...
grey: extent of image to be processed white: padded area (in my case filled with 2.0's) red shades: extent of kernel dark red: effective neighbourhoud light red: part of neighbourhood to be ignored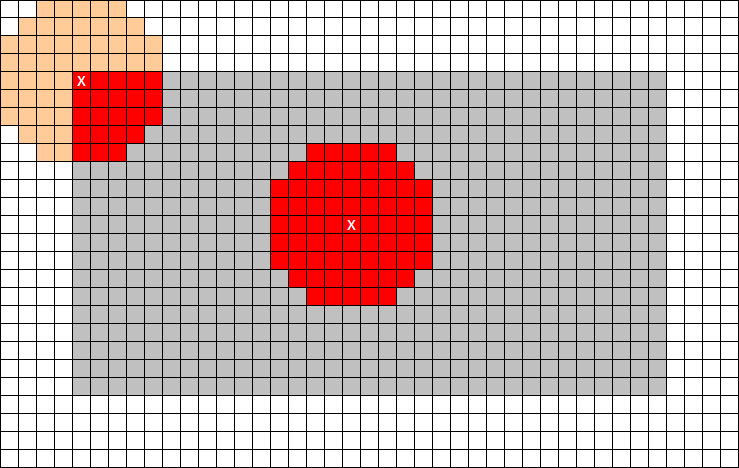
Как можно изменить этот довольно прагматичный кусок кода для повышения производительности вычислений?
Спасибо заранее!